Distanz-Matrix
Die Elemente einer Distanz-Matrix enthalten Werte, die die kürzeste Distanz zwischen den betrachteten Atomen spezifiziert. Distanzen können entweder als geometrische (in Å) oder als topologische Abstände (Anzahl der Bindungen) ausgedrückt werden.
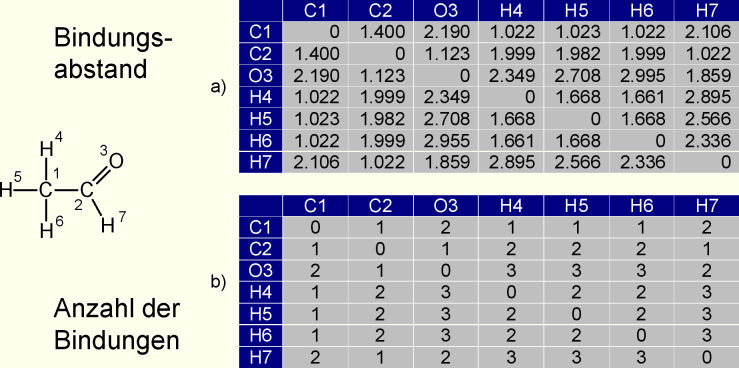
Distanz-Matrizen von Ethanal: a) geometrische
Distanzen in Å und b) topologische Abstände in Bindungslängen
© Prof. Dr. J. Gasteiger, Dr. Th. Engel, CCC Univ. Erlangen, Thu Dec 18 14:53:53 2003 GMT
 BMBF-Leitprojekt Vernetztes Studium - Chemie BMBF-Leitprojekt Vernetztes Studium - Chemie
|


 BMBF-Leitprojekt Vernetztes Studium - Chemie
BMBF-Leitprojekt Vernetztes Studium - Chemie
