R/S-Stereochemie im Molfile
Ein Struktureditor kann eine chemische Verbindung zeichnen und
z.B. im Molfile-Format abspeichern. Obwohl es möglich ist,
in diese Zeichnung stereochemische Eigenschaften als Keil oder gestrichelte
Bindung einzubringen, bzw. das Stereozentrum oder die Stereogruppe
mit dessen Auszeichnungen (R/S oder E/Z) zuordnen,
stellt die Bindungsliste (CT) des Molfiles nur die Konstitution
(Topologie) des Moleküls dar.
Zusätzliche Stereoinformation muss von der graphischen Darstellung abgeleitet und in Stereodeskriptoren codiert werden (s. Permutationsdeskriptoren). Die Stereodeskriptoren werden dann in entsprechende Felder der Bindungsliste geschrieben.
Der stereochemische Deskriptor der im Molfile zu finden ist, basiert
auf der geordneten Liste von Petrarca, Lynch und Rush (PLR). Wipke
und Dyott entwickelten dieses Konzept im SEMA- (Stereochemically
Extended Morgan Algorithm) Projekt in den frühen 70er weiter.
Wie der Name schon verrät, werden die Atome um das Stereozentrum
nach dem Morgan Algorithmus nummeriert. Mit Kenntnis der Priorität
der Liganden und der Anordnung in der PLR-Liste, kann der Stereodeskriptor
bestimmt werden.
Da die Stereochemie nicht immer bestimmt werden kann bzw. manchmal
unbekannt ist, muss der Deskriptor mehr als nur die beiden Werte
für R und S beinhalten. Dieses Problem wurde
mit dem "Paritäts-Wert" gelöst.
Der Paritäts-Wert kann berechnet werden, indem er mit der geordneten Liste verglichen wird. Hierbei werden die durch den Morgan-Algorithmus erhaltenen Indizes so lange vertauscht bis sie in steigender Ordnung sind. Um den Paritäts-Wert nun zuordnen zu können, ist die Anzahl der Permutationen wichtig. Ist die Anzahl ungeradzahlig, ist der Paritäts-Wert 1, ist er geradzahlig, wird der Wert 2. Weiterhin kann der Paritäts-Wert eine 0 bekommen, falls keine Stereochemie am Stereozentrum definiert ist. Ist die Konfiguration unbekannt ist der Wert 3.
Bestimmung des Paritäts-Wertes:
- eine vorgegebene, willkürlich bezifferte Struktur wird
mit Hilfe des Morgan-Algorithmus kanonisiert (im Beispiel sind
nur am Stereozentrum die Morgan-Nummern aufgeführt)
- dann werden die Morgan-Nummern der dem Stereozentrum benachbarten
Atome aufgelistet, beginnend mit der kleinsten Zahl. Die weiteren
drei Zahlen folgen nach dem Uhrzeigersinn, wobei die Blickrichtung
vom Nachbaratom mit der kleinsten Zahl zum Stereozentrum geht.
- der Paritäts-Wert wird durch die Anzahl der Permutationen
bestimmt, die nötig sind, um die Indizes in steigender Ordnung
zu bringen (Anzahl ungeradzahlig=1, geradezahlig=2).
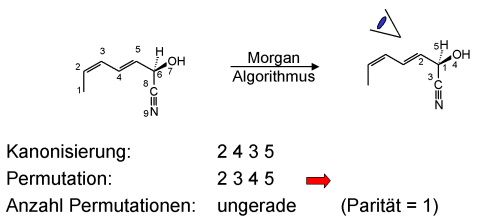
Bestimmung der Parität
© Prof. Dr. J. Gasteiger, Dr. Th. Engel, CCC Univ. Erlangen, Thu Dec 18 14:53:54 2003 GMT
 BMBF-Leitprojekt Vernetztes Studium - Chemie BMBF-Leitprojekt Vernetztes Studium - Chemie
|


 BMBF-Leitprojekt Vernetztes Studium - Chemie
BMBF-Leitprojekt Vernetztes Studium - Chemie
