Interne Koordinaten - Z-Matrix
Interne Koordinaten beschreiben die räumliche Anordnung der
Atome relativ zueinander unter Benutzung von Bindungslängen,
Bindungswinkel und Torsionswinkel. Eine recht gebräuchliche
Art, interne Koordinaten anzugeben stellt die Z-Matrix dar.
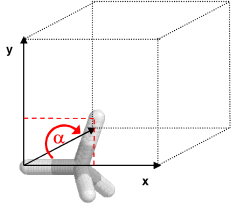
Internes Koordinatensystem
Um eine Z-Matrix aufzustellen, sind einige Regeln zu befolgen.
Jede Zeile in der Z-Matrix stellt ein Atom des Moleküls dar.
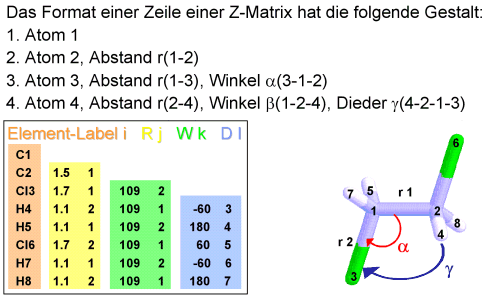
Z-Matrix von 1,2-Dichlorethan
Im Beispiel ist in der ersten Zeile das Atom 1 als C1 definiert,
dieses Kohlenstoffatom liegt im Ursprung des Koordinatensystems.
Das zweite Atom, C2, hat einen Abstand von 1.5 Å (2. Spalte)
vom Atom 1 (3. Spalte) und sollte immer auf der x-Hauptachse liegen.
Das dritte Atom, das Chlor-Atom Cl3, muss in der xy-Ebene liegen.
Es hat einen Abstand von 1.7 Å von Atom 1, und der Winkel
a zwischen den Atomen 3-1-2 beträgt
109° (4. und 5. Spalte). Ab der vierten Zeile der Z-Matrix wird
die genaue Lage jedes Atoms, neben dem Abstand r und dem
Winkel a, mit dem Torsions- oder Dieder-Winkel
g beschrieben (6. und 7. Spalte). Dieser
Winkel wird z.B. zwischen der xy-Ebene und einem Atom außerhalb
dieser Ebene ausgebildet (z.B. 4-2-1-3).
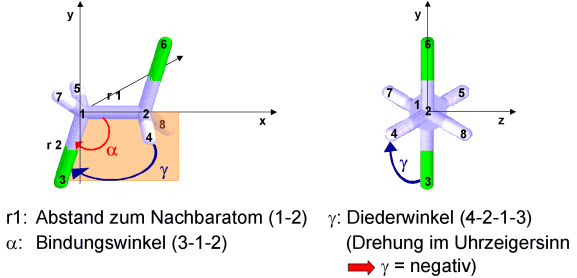
Interne Koordinaten von 1,2-Dichlorethan: Bindungslängen
r1 und r2, Bindungswinkel a und Torsionswinkel
g
Mit Ausnahme der ersten drei Atome, wird jedes Atom durch einen
Satz aus drei internen Koordinaten beschrieben: eine Distanz zu
einem vorher definierten Atom, einen Bindungswinkel der aus dem
Atom mit zwei vorigen Atomen gebildet wird, und einen Torsionswinkel
des Atoms mit drei vorigen Atomen. Insgesamt werden 3N-6 interne
Koordinaten zur genauen Beschreibung einer chemischen Struktur im
3D-Raum benötigt, wobei N die Anzahl der Atome im Molekül
ist. Die Anzahl der internen Koordinaten (3N-6) entspricht damit
auch der Anzahl der Freiheitsgrade eines Moleküls.
© Prof. Dr. J. Gasteiger, Dr. Th. Engel, CCC Univ. Erlangen, Thu Dec 18 14:53:54 2003 GMT
 BMBF-Leitprojekt Vernetztes Studium - Chemie BMBF-Leitprojekt Vernetztes Studium - Chemie
|


 BMBF-Leitprojekt Vernetztes Studium - Chemie
BMBF-Leitprojekt Vernetztes Studium - Chemie


