Strukturcodierung
Ein grundlegender Aspekt, in dem sich die hier vorgestellte Methode der Spektrenvorhersage mit Hilfe eines neuronalen Netzes von anderen Vorhersage- oder Berechnungsmethoden unterscheidet, ist, daß sie prinzipiell auf beliebige Moleküle anwendbar ist. Die Vorhersagegeschwindigkeit und Qualität ist unabhängig von der Molekülgröße. Da das neuronale Netz an Beispielen lernt, ist die Vorhersagequalität abhängig von der Qualität der zugrundeliegenden Datenbasis und der Repräsentation der jeweiligen Anfragestruktur durch die Datenbasis. Ein Schlüsselschritt dieser Methode ist dabei die Art der Strukturbeschreibung, mit der Molekülstrukturen dem neuronalen Netz präsentiert werden.
Neuronale Netze die zur Simulation von IR-Spektren verwendet werden und statistische Methoden benötigen als Eingabeformat einen Datenvektor mit konstanter Länge. Da sowohl die Zahl der Atome und deshalb die Zahl der kartesischen Koordinaten, als auch die physikochemischen Werte von Molekül zu Molekül verschieden sind, muss ein Weg gefunden werden, diese Daten in eine eindimensionale mathematische Funktion zu konvertieren. Diese Prozedur nennt man Strukturcodierung.
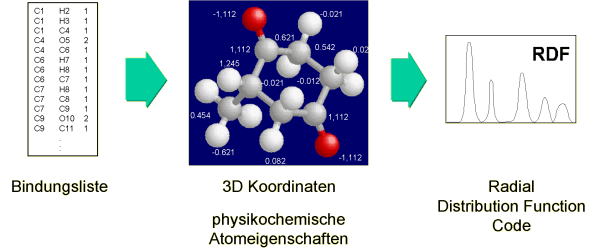
Nachdem aus der Bindungsliste eine 3D Struktur mit physikochemischen Eigenschaften berechnet worden ist, erfolgt die Strukturcodierung durch den RDF Code
Zur Simulation von IR-Spektren wurden zwei Wege zur Codierung einer 3D-Struktur entwickelt: die radiale Verteilungsfunktion und der 3D-MoRSE-Code.
© Prof. Dr. J. Gasteiger, Dr. Th. Engel, CCC Univ. Erlangen, Wed Jun 9 12:55:24 2004 GMT
 BMBF-Leitprojekt Vernetztes Studium - Chemie BMBF-Leitprojekt Vernetztes Studium - Chemie
|


 BMBF-Leitprojekt Vernetztes Studium - Chemie
BMBF-Leitprojekt Vernetztes Studium - Chemie
