Das Pdb-Format
Das Pdb-Format hat seinen Namen von der amerikanischen Brookhaven Protein Database.
Seit 1998 bei Research Collaboratory for Structural Bioinformatics (RCSB).
Die Proteindatenbank archiviert seit 1971 Kristallstrukturen (3D-Strukturen)
von biologischen Molekülen. Zusätzlich zu den Atomkoordinaten sind
in den Moleküldateien Angaben (Remarks) zur Struktur (kristallographische
Daten, Primär-, Sekundärstrukturen), experimentelle Daten (NMR, etc.)
oder Literaturangaben enthalten. Durch diese differenzierten Informationen erlaubt
die Datenbank eine individuelle Recherche von Daten.
Auch die pdb-Datei ist eine tabellarische Textdatei (ASCII) mit einer Zeilenlänge
von 80 Zeichen. Im Gegensatz zum Molfile sind viele Zusatzinformationen enthalten.
Um die zusätzlichen Daten zu klassifizieren und zu identifizieren, werden
bestimmte Schlüsselwörter "Keywords" an den Zeilenanfang gestellt.
Wie im Molfile sind die eigentlichen Strukturdaten in einer CT enthalten. Im
Atomblock stehen die Atomnamen und die xyz-Koordinaten der verschiedenen Atome.
Der Bindungsblock gibt Auskunft über die Verknüpfung der Atome untereinander.
Was nicht angegeben wird, ist die Wertigkeit der Bindung, d.h. Angaben über
Mehrfachbindungen.
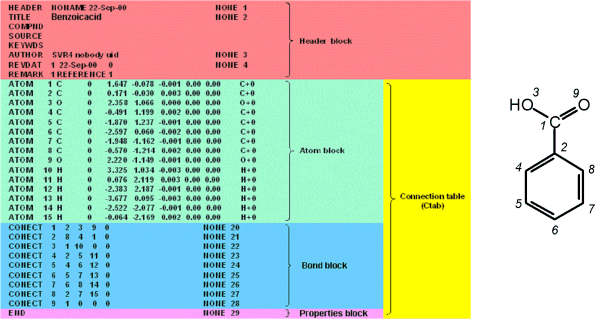
Prinzipieller Aufbau eines PDB-Files einer chemischen Verbindung (Benzoesäure)
Tutorien:
© Prof. Dr. J. Gasteiger, Dr. Th. Engel, CCC Univ. Erlangen, Wed Apr 7 12:05:55 2004 GMT
 BMBF-Leitprojekt Vernetztes Studium - Chemie BMBF-Leitprojekt Vernetztes Studium - Chemie
|


 BMBF-Leitprojekt Vernetztes Studium - Chemie
BMBF-Leitprojekt Vernetztes Studium - Chemie
