Atom-by-Atom Match
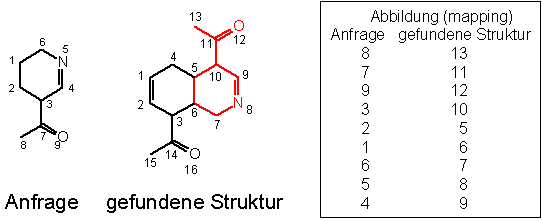
Um zu bestimmen, ob eine Struktur die Substruktur der Suchanfrage enthält, muß ein Algorithmus angewendet werden, der alle Atome und Bindungen beider Strukturen aufeinander abbildet und vergleicht.
Dieser Algorithmus ist abhängig von der Anzahl der Atome und Bindungen. Um Fehler möglichst früh auszuschließen, werden vorzugsweise die Atome mit kleiner Anzahl (also hier die Heteroatome N und O) als Startatome gewählt. Daneben wird die Atomumgebung betrachtet. Atom 3 ist in der Substruktur Teil eines Heterocyclus, im Zielmolekül ist dies für Atom 3 nicht der Fall.
© Prof. Dr. J. Gasteiger, Dr. A. Schunk, Dr. Th. Engel, CCC Univ. Erlangen,
Wed Jun 9 13:49:45 2004 GMT
 BMBF-Leitprojekt Vernetztes Studium - Chemie BMBF-Leitprojekt Vernetztes Studium - Chemie
|


 BMBF-Leitprojekt Vernetztes Studium - Chemie
BMBF-Leitprojekt Vernetztes Studium - Chemie