Optimierung des Backtracking Algorithmus
Zur Verkürzung der Rechenzeiten gibt es unterschiedliche Ansätze, Substruktursuchen nach einem Backtracking Algorithmus zu optimieren.
1. Verbesserung der Hard- und Software
Neben schnelleren Computersystemen kann eine Beschleunigung auch durch Verwendung paralleler Rechner-Architekturen erreicht werden.
2. Anwendung von heuristischen Methoden
Dabei werden vor allem lokale Eigenschaften der Atome berücksichtigt. (Atomtyp, Anzahl der Bindungen, Bindungsordnung, Ringgröße) H-Atome können (zunächst) unterdrückt werden; die Suche kann an Atomen möglichst niedriger Konnektivität begonnen werden.
3. Preprocessing
Die Strukturen in der Datenbank werden vorab unabhängig von Suchanfragen auf mögliche Substrukturen (Schlüssel) überprüft. Als Ergebnis wird ein Bit-String konstruiert, wobei jedes Bit die An- oder Abwesenheit des jeweiligen Schlüssels (also einer definierten Substruktur) repräsentiert. Bei der Substruktursuche kann dann bereits durch Abgleich mit den Schlüsseln (Screening) ein Großteil der Strukturen eliminiert werden.
4. Partitionierung
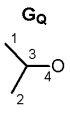
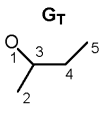
Die Atome werden nach Element und Konnektivität in Klassen eingeteilt. (Sussenguth, 1965) Ist die entsprechende Klasse in der Struktur nicht vorhanden, bleibt sie auch bei der Suche unberücksichtigt.
zum Beispiel:
| |
|
|
|
Klassenbeschreibung
|
Atome von GQ
|
Atome von GT
|
C-Atom mit 1 Einfach-Bindung (Klasse I)
|
1, 2
|
2, 5
|
C-Atom mit 2 Einfach-Bindungen (Klasse II)
|
|
4
|
C-Atom mit 3 Einfach-Bindungen (Klasse III)
|
3
|
3
|
O-Atom mit 1 Einfach-Bindung (Klasse IV)
|
4
|
1
|
Nur wenn alle definierten Klassen der Suchanfrage (von GQ) auch im Zielmolekül (GT) vorhanden sind, ist ein Isomorphismus möglich. Im Beispiel werden von Anfragemolekül 3 Klassen (1,3,4) und vom Zielmolekül alle 4 Klassen belegt, d.h. es ist prinzipiell Isomorphismus möglich.
Durch mehrmalige Partitionierung und Relaxation (Berücksichtigung der Nachbarn) kann die Laufzeit auf polynomiale Ordnung reduziert werden.
© Prof. Dr. J. Gasteiger, Dr. A. Schunk, Dr. Th. Engel, CCC Univ. Erlangen,
Wed Jun 9 13:49:45 2004 GMT
 BMBF-Leitprojekt Vernetztes Studium - Chemie BMBF-Leitprojekt Vernetztes Studium - Chemie
|


 BMBF-Leitprojekt Vernetztes Studium - Chemie
BMBF-Leitprojekt Vernetztes Studium - Chemie