JME Molecular Editor
Der JME Editor von Peter Ertl, ist ein Java Programm, welches erlaubt, Moleküle
und Reaktionen direkt in einer Internetseite zu zeichnen, zu editieren und anzuzeigen.
Es kann aber auch als Stand-alone Anwendung genutzt werden. Der Editor war ursprünglich
für ein lokales, Web-basiertes Chemoinformatik-System entwickelt worden,
ist aber aufgrund großer Nachfrage jedem zugänglich gemacht worden.
JME ist mit mehr als tausend Installationen momentan wahrscheinlich das populärste
Web-Moleküleingabe-System. Das JME Applet wird u.a. für Moleküldatenbanken,
Eigenschaftsvorhersage-Services, verschiedene Chemoinformatik Tools (z.B. 3D-Strukturgeneratoren
oder Molekülorbital Visualisierungen) und interaktive Seiten zur Lehre
genutzt.
Die Grundfunktion des JME Editors ist die Erzeugung und Modifikation von Molekülen und Reaktionen direkt in der Web-Seite. Der Editor hat alle standardmäßigen Zeichen- und Editiermöglichkeiten. Diese enthalten ein großes Set an Tastatur-Shortcuts zum einfachen Hinzufügen von Strukturfragmenten.
SMILES code, MDL Molfile oder ein JME eigenes, kompaktes Format (einzeilige Representation eines Moleküls oder einer Reaktion inkl. 2D Koordinaten) können von erzeugten Molekülen generiert werden. Der erzeugt SMILES-code ist unabhängig von der Art wie das Molekül gezeichnet worden ist (Unique SMILES). Eine Erweiterung von JME, in Kooperation mit H. Rzepa und P. Murray-Rust, erlaubt auch die Ausgabe der Moleküle im CML (Chemical Markup Language) Format.
Der JME kann mit Hilfe von einfachen HTML Formular-Elementen als Eingabe-Tool für Strukturdatenbank-Abfragen dienen, welches komplexe Strukturanfragen erlaubt, wie z.B. eine 3D Pharmakophorsuche in der NCI Datenbank. Reaktionen werden durch Erzeugung von "reaction SMILES" und SMIRKS unterstützt.
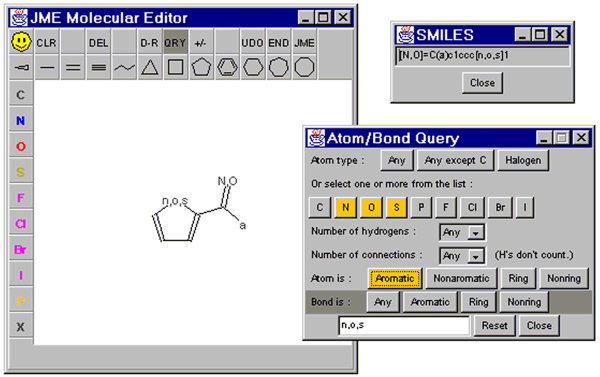
Mit dem JME-Applet sind neben einfachen Molekülzeichnungen auch Substruktur-Anfragen
möglich, die Teil eines komplexeren Moleküls darstellen
Das Applet kann im Darstellungsmodus (ohne Editierleiste) genutzt werden um Moleküle zu visualisieren.
Außerdem ist eine Kommunikation mit anderen Elementen einer Webseite (andere Applets oder JavaScript Objekte) über die "public function" möglich.
http://www.xml-cml.org/downloads/jme/
© Prof. Dr. J. Gasteiger, Dr. Th. Engel, CCC Univ. Erlangen, Wed Apr 7 12:05:54 2004 GMT
 BMBF-Leitprojekt Vernetztes Studium - Chemie BMBF-Leitprojekt Vernetztes Studium - Chemie
|


 BMBF-Leitprojekt Vernetztes Studium - Chemie
BMBF-Leitprojekt Vernetztes Studium - Chemie
