Molekül-Viewer
Während mit Moleküleditoren auf einfache Art und Weise chemische Strukturen gezeichnet werden können, dienen Viewer der räumlichen Darstellung dieser Strukturen.
Während ein Editor hauptsächlich zur Erzeugung eines Molekül-Strukturcodes eingesetzt wird, wie z.B. SMILES oder Bindungslisten (Connection Tables) in Molfiles, benutzen Molekülbetrachtungs-Programme (Viewer) diesen Code um die Strukturen in geeigneter Weise zu visualisieren.
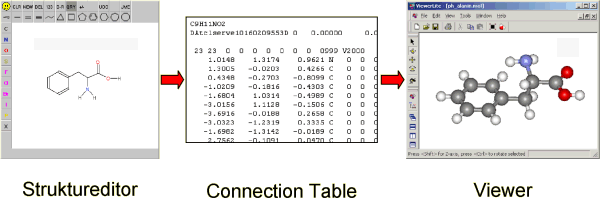
Struktureditoren (links) erzeugen Strukturcodes (Mitte), welche in Molekülbetrachtungs-Programmen (rechts) in ansprechender Weise visualisiert werden können
Um eine oder mehrere Molekülstrukturen räumlich darzustellen, werden Strukturdateien mit 3D Informationen (z.B. pdb, xyz, cif, mol, etc.) benötigt. Wenn Strukturen direkt von einem Struktureditor kommen, enthalten die Dateien normalerweise keine 3D Daten. Dies kann nur mit 3D Strukturgeneratoren oder Kraftfeld-Berechnungen, etc., erfolgen. Danach können die 3D Strukturen in verschiedenen Darstellungsmodi wie z.B. Draht-, Kugelstab- oder Kalotten-Modell repräsentiert werden. Proteine werden meist als Bandmodell visualisiert, wobei die Sekundärstrukturen (Helices, Faltblätter) besonders hervorgehoben und die Tertiärstrukturen damit sichtbar werden. Weitere Features ermöglichen es, die Atome entsprechend den Untereinheiten, Temperaturfaktoren oder Kettentypen zu färben. Bei allen Operationen kann das Molekül vom Nutzer interaktiv bewegt, rotiert oder skaliert werden.
Insbesondere molekularbiologische Datenbank-Provider bieten verschiedene Viewer z.B. Swiss-PdbViewer (SPDBV 3.7; http://www.expasy.org/swissmod) mit konventionellen Optionen an. Dies sind spezialisierte interaktive Molekülgrafik Programme zum Betrachten und Analysieren von Proteinen und Nukleinsäuren.
© Prof. Dr. J. Gasteiger, Dr. Th. Engel, CCC Univ. Erlangen, Wed Apr 7 12:05:54 2004 GMT
 BMBF-Leitprojekt Vernetztes Studium - Chemie BMBF-Leitprojekt Vernetztes Studium - Chemie
|


 BMBF-Leitprojekt Vernetztes Studium - Chemie
BMBF-Leitprojekt Vernetztes Studium - Chemie
