Stand-alone Anwendungen von Viewer
Einige Stand-alone Struktureditor-Programme werden durch eine integrierte 3D-Visualisierungs-Anwendung ergänzt (ChemWindow Þ SymApps, ChemSketch Þ ACD/3D Viewer, ChemDraw Þ Chem3D). Diese relativ einfachen Viewer generieren vorwiegend die 3D Geometrien mithilfe Kraftfeld-Berechnungen. Grundlegende Visualisierungs- und Manipulierungs-Möglichkeiten werden ebenfalls zur Verfügung gestellt. Die Molekülmodelle können in unterschiedliche Darstellungsstile, Farben, Schattierungen etc. visualisiert werden und sind auf dem Bildschirm skalierbar, bewegbar und rotierbar.
ISIS/Draw 2.4
ISIS/Draw hat kein eigenes Molekülvisualisierungs Tool. Das "rotate tool" tauscht nur das 2D in ein 3D "rotate tool", welches dann die 2D Struktur in drei Dimensionen bewegen lässt. Um die gezeichneten Strukturen dreidimensional zu visualisieren, ist es notwendig, die Zeichnung in einen Viewer wie z.B. ACD/3D zu kopieren.
ACD/3D Viewer V 5.07
Dieses Viewer-Modul ist komplett in das ACD/Chemsketch Zeichenprogramm integriert
und erlaubt somit schnell, durch Mausklick, eine 3D Darstellung zu erhalten.
Die 3D Moleküle können dann bewegt, rotiert, etc. (aber nicht vergrößert)
und in verschiedenen Stilen, Fonts und Farben dargestellt werden. Außerdem
kann die Struktur mit Hilfe von Kraftfeldberechungen (CHARMM-Typ) optimiert
werden. Bindungslängen, Bindungswinkel und Torsionswinkel können gemessen
werden. Die erzeugte 3D Struktur-Darstellung kann nur im ACD-eigenen Format
abgespeichert, oder in Grafik-Anwendungsprogramme kopiert werden.
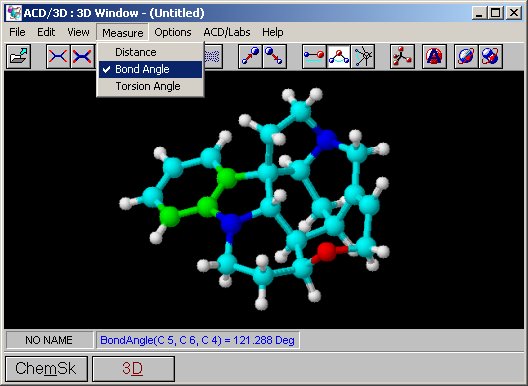
User-interface vom ACD/3D Viewer; Der Bindungswinkel zwischen den drei grün markierten Atomen wird in der unteren Leiste angezeigt
Chem3D UltraV 6.0
Chem3D ist mehr als ein einfacher Molekülviewer. Das eigenständige Softwaremodul des ChemOffice Paketes stellt auch einfache Molekülzeichen-Tools zur Verfügung um Strukturen zu erzeugen. Diese werden dann v.a. als Eingabe für Molecular Modelling Anwendungen genutzt, die als Ergebnis 3D Strukturen ergeben, die wiederum visualisiert werden können.
SymApps V. 5.0
SymApps konvertiert mit Hilfe von modifizierten MM2-Kraftfeldberechnungen, 2D Strukturen vom ChemWindow Zeichenprogramm in 3D Darstellungen. Neben grundlegenden Visualisierungstools wie z.B. Darstellungsstil, perspektivische Ansichten und Lichtquellenanordnungen, stellt das Modul zusätzliche Berechnungsmöglichkeiten für Bindungslängen und -winkel etc. zur Verfügung. Außerdem können Punktgruppen und Charaktertafeln bestimmt werden. Animierte Rotationsbewegungen und Symmetrieoperationen können erzeugt und als Movie-Datei (*.avi) gespeichert werden.
User-interface von SymApps
© Prof. Dr. J. Gasteiger, Dr. Th. Engel, CCC Univ. Erlangen, Wed Apr 7 12:05:54 2004 GMT
 BMBF-Leitprojekt Vernetztes Studium - Chemie BMBF-Leitprojekt Vernetztes Studium - Chemie
| 

 BMBF-Leitprojekt Vernetztes Studium - Chemie
BMBF-Leitprojekt Vernetztes Studium - Chemie

