Fingerprints
Ein Fingerabdruck einer chemischen Struktur versucht ein Molekül anhand spezifischer Charakteristika zu identifizieren. Im Strukturschlüssel lassen sich Fragmente chemischer Strukturen codieren. Hierbei werden Fragmente in einer Struktur als Abfolge von 0 und 1 dargestellt (Bitstring). 0 bedeutet daß das Fragment nicht in der Struktur vorhanden ist, bei 1 ist das Fragment mindestens einmal vertreten. Typische Längen von Fingerprints sind 150-2500 Bits. bei gleicher Größe von Bitlänge und Anzahl an Fragmenten in der Bibliothek ist eine 1:1 Korrelation zwischen Fragment und Bit im Fingerprint möglich. Falls eine Struktur nur wenige definierte Fragmente besitzt, werden auch nur wenige Bits gesetzt.
|
In diesem Beispiel stehen in der Bibliothek
nur -NH2 und -C=O als Fragmente zur Verfügung.
Ein Binärcode könnte somit folgendermassen ausschauen:
|
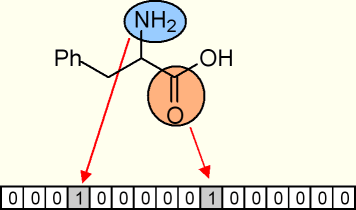 |
"Hashed Fingerprints"
Bei diesem Verfahren werden alle Bindungswege im Molekül von
einem Atom bis zu mehreren Bindungslängen (z.B. sieben) abgelaufen
und virtuell "zerhackt = hashed". Hierbei erhält
man Information über die Substrukturen des Moleküls aber
auch über dessen internen Beziehungen. Jedes erhaltene Fragment
wird im Bitstring mit 1 ausgewiesen. Der so erhaltene Fingerprint
kann jedoch aufgrund des Algorithmus Kollisionseinträge enthalten.
Der Vorteil gegenüber dem "normalen" fragmentbasierten Fingerprint
ist, dass keine Fragmente vordefiniert werden müssen und somit
eine bessere Beschreibung der Struktur erhalten wird. Allerdings
ist durch den Wegfall der Fragmentbibliothek keine direkte Korrelation
eines Biteintrages mit einer Substruktur möglich.
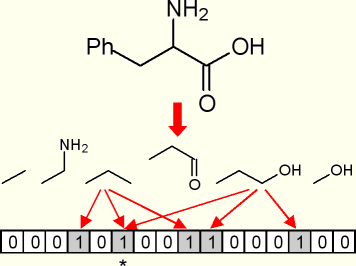 |
Dieser Fingerprint wurde durch Hashing
erhalten, wobei in der Abbildung nur ein Teil aller Substrukturen
aufgeführt ist. Das Sternchen markiert eine Adresskollision
des Bitstrings, die aufgrund des Algorithmus entstanden ist.
|
© Prof. Dr. J. Gasteiger, Dr. Th. Engel, CCC Univ. Erlangen, Thu Dec 18 14:53:53 2003 GMT
 BMBF-Leitprojekt Vernetztes Studium - Chemie BMBF-Leitprojekt Vernetztes Studium - Chemie
|


 BMBF-Leitprojekt Vernetztes Studium - Chemie
BMBF-Leitprojekt Vernetztes Studium - Chemie

