Kanonisierung
In Strukturdatenbanken können Verbindungen mit bereits vorhandenen Einträgen verglichen werden. Dazu muß der Informationscode, der ein Molekül beschreibt, eineindeutig sein. Dieser kann aus der Anzahl der in einem Molekül vorhandenen Atome, der Atomart (z.B. Kohlenstoff, Sauerstoff), der durch Bindungen verknüpften Atome und der Art der Bindung (z.B. Einfach-, Doppelbindung) abgeleitet werden. Würden ausschließlich die Atome eines Moleküls betrachtet, welche willkürlich nummeriert bzw. bezeichnet werden können, wären prinzipiell bei n Atomen n! Beschreibungsmöglichkeiten dieser einen Struktur gegeben, und somit vorerst keine eineindeutige Codierung möglich.
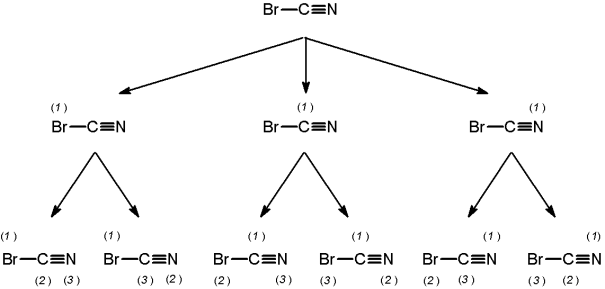
Ein Molekül mit nur drei Atome, wie zum Beispiel CNBr, kann
durch 3! also 6 differenzierte Atomnummerierungen gekennzeichnet
und somit in 6 unterschiedliche Connection Tables beschrieben werden.
Mit Hilfe eines "Stammbaumes" kann die unterschiedliche Aufstellung
der Numerierung verfolgt werden:
Sechs unterschiedliche Nummerierungsmöglichkeiten
der Atome im CNBr
Ziel der Kanonisierung ist es nun aus der Vielzahl an Atom-Nummerierungen eine als Standard herauszunehmen um daraus einen eineindeutigen Code für eine CT oder eine Bindungsmatrix ableiten zu können. Solch eine einzigartige, reproduzierbare Nummerierung der Atome kann nur durch einige Regeln erhalten werden. Dazu stehen verschiedene Möglichkeiten zur Verfügung:
- Matrizen-Methode, Eigenvektoren (dieses Verfahren hat sich nicht durchgesetzt)
- Morgan-Algorithmus
© Prof. Dr. J. Gasteiger, Dr. Th. Engel, CCC Univ. Erlangen, Thu Dec 18 14:53:53 2003 GMT
 BMBF-Leitprojekt Vernetztes Studium - Chemie BMBF-Leitprojekt Vernetztes Studium - Chemie
|


 BMBF-Leitprojekt Vernetztes Studium - Chemie
BMBF-Leitprojekt Vernetztes Studium - Chemie