Distanz- und Ähnlichkeitsmaße
Die Ähnlichkeit zwischen Verbindungen wird im Sinne von Abstandsmaßen dst zwischen zwei unterschiedlichen Objekten s und t bewertet. Die Objekte s und t werden beschrieben durch die Vektoren xs=(xs1, xs2, ... xsm) und xt=(xts, xt2, ..., xtm) wobei m die Anzahl an realen Variablen angibt, xsj und xtj sind jeweils das j-te Element des entsprechenden Vektors. Für die Berechnung der Distanz bzw. der Ähnlichkeit zweier Verbindungen, sollten die Variablen xj vergleichbare Größe haben. Andernfalls muß eine Skalierung oder Normalisierung durchgeführt werden.
Die zwei bedeutendsten Distanzmaße sind:
| Euklidsche Distanz: |
 |
| Manhatten Distanz: |
 |
Ein Ähnlichkeitsmaß 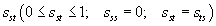 kann aus dem Distanzmaß dst durch die folgende Gleichung berechnet werden, wobei dst das Distanzmaß für die Objekte s und t ist und dmax die maximale Distanz zwischen einem Objektpaar des Datensatzes. kann aus dem Distanzmaß dst durch die folgende Gleichung berechnet werden, wobei dst das Distanzmaß für die Objekte s und t ist und dmax die maximale Distanz zwischen einem Objektpaar des Datensatzes.

Ein weiteres, häufig benutztes Ähnlichkeitsmaß, ist der Kosinuskoeffizient, der sich aus dem Kosinus des Winkels von zwei Vektoren und deren Skalarprodukt berechnen läßt.
Die Berechnung der Distanzmaße für zwei Objekte s und t, dargestellt durch binäre Deskriptoren xs und xt mit m binären Werten, basiert auf der Häufigkeit der gemeinsamen und unterschiedlichen Komponenten. Zu diesem Zweck werden die Häufigkeiten a,b,c und d wie folgt definiert:
a: Anzahl der Komponenten mit xsj=1 und xtj=1
b: Anzahl der Komponenten mit xsj=1 und xtj=0
c: Anzahl der Komponenten mit xsj=0 und xtj=1
d: Anzahl der Komponenten mit xsj=0 und xtj=0
Die Häufigkeiten a und d spiegeln die Ähnlichkeit zwischen zwei Objekten s und t wider, wohingegen b und c Information zur Diversität beitragen.
| Hamming Distanz: |
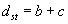 |
| Tanimoto-Koeffizient: |
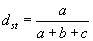 |
| Kosinus-Koeffizient: |
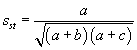 |
Sind die binären Deskriptoren der Objekte s und t Substruktur-Schlüssel, gibt die Hamming Distanz die Anzahl der unterschiedlichen Substrukturen in s und t an (Komponenten die sowohl in s als auch in t 1 sind, nicht aber in beiden). Der Tanimnoto-Koeffizient ist andererseits ein Maß für die Anzahl an Substrukturen, die s und t gemeinsam haben (d.h. die Häufigkeit a), bezogen auf die Gesamtzahl der Substrukturen, die sie teilen können (gegeben durch die Anzahl der Komponenten die sowohl in s oder in t 1 sind).
© Prof. Dr. J. Gasteiger, Dr. Th. Engel, CCC Univ.
Erlangen, Wed Jun 9 12:55:24 2004 GMT
 BMBF-Leitprojekt
Vernetztes Studium - Chemie BMBF-Leitprojekt
Vernetztes Studium - Chemie
|


 BMBF-Leitprojekt
Vernetztes Studium - Chemie
BMBF-Leitprojekt
Vernetztes Studium - Chemie