Feature Trees
Das Konzept der Feature Trees (Ftrees) wurde von Rarey und Dixon eingeführt und basiert auf einem Vergleich von Molekülen unter Berücksichtigung der relativen Anordnung potentieller Interaktionsgruppen in den Molekülen. Die Ähnlichkeit zweier Moleküle kann somit durch Molekülprofile und einer groben Zuordnung (Mapping) berechnet werden.
Im Gegensatz zu den linearen Deskriptoren (Fingerprint, Bitstring, etc.) wird beim Feature Tree ein Molekül durch eine Baumstruktur dargestellt (nicht-linearer Moleküldeskriptor). Die Knoten des Baums repräsentieren ganze Fragmente des Moleküls, und die Kanten werden analog zu den Bindungen des Molekülgraphen gezogen. Jeder Knoten trägt somit Informationen über die sterischen und physikochemischen Eigenschaften (Atomtypen) des repräsentierten Molekülfragments.
Werden nun zwei Moleküle miteinander verglichen, besteht die Aufgabe darin, zunächst eine Zuordnung der Fragmente der Moleküle zu finden, die bei einer Bindung an ein Protein die gleichen Regionen des aktiven Zentrums belegen könnten. Auf der Basis dieser Zuordnung kann dann ein Ähnlichkeitswert ermittelt werden der die Topologie des Moleküls berücksichtigt.
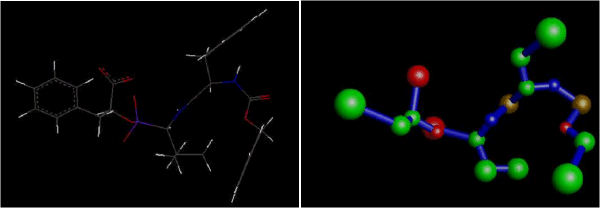
Ein Wirkstoffmolekül (links) und der zugehörige Feature Tree (rechts). Die Knoten des Feature Trees sind nach den chemischen Eigenschaften der repräsentierten Molekülgruppen gefärbt: H-Donor (blau); H-Akzeptor (rot); Phenylring (grün); CH3, Amid (grün); hydrophob (grün);
(http://cartan.gmd.de/ftrees/)
Neben dem Vergleich von einzelnen Molekülpaaren eignet sich der Feature-Tree Deskriptor insbesondere zur Suche in kombinatorischen Bibliotheken.
M. Rarey, J. S. Dixon, J. Comput.-Aided Mol. Design 1998, 12, 471-490
© Prof. Dr. J. Gasteiger, Dr. Th. Engel, CCC Univ.
Erlangen, Wed Jun 9 12:55:24 2004 GMT
 BMBF-Leitprojekt
Vernetztes Studium - Chemie BMBF-Leitprojekt
Vernetztes Studium - Chemie
| 

 BMBF-Leitprojekt
Vernetztes Studium - Chemie
BMBF-Leitprojekt
Vernetztes Studium - Chemie
