Radiale Verteilungsfunktion
Steinhauer und Gasteiger entwickelten einen neuen 3D Deskriptor, der auf der Idee der radialen Verteilungsfunktion (radial distribution function = RDF) aufbaut. Dieser ist im Allgemeinen in der Physik und der physikalischen Chemie und insbesondere in der Röntgenstrukturaufklärung bestens bekannt. Die radiale Verteilungsfunktion ist nahe verwandt mit dem 3D MoRSE-Code.
Der RDF Code wird berechnet nach
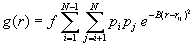
wobei f ein Skalierungsfaktor ist, N ist die Anzahl der Atome im Molekül, pi und pj sind Eigenschaften der Atome i und j, B ist ein Glättungsparameter und rij ist die Distanz zwischen den Atomen i und j; g(r) wird normalerweise als diskrete Funktion berechnet aus einer Anzahl diskreter Punkte innerhalb definierter Intervalle.
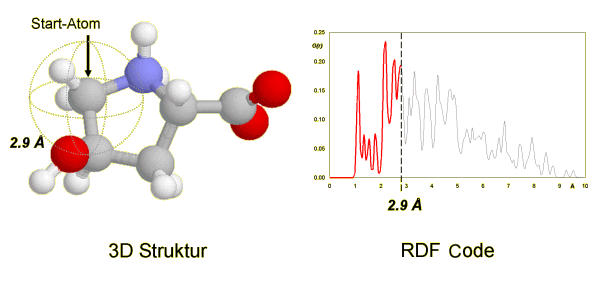
RDF-Codierungsprozedur einer 3D Struktur
Eine etwas vereinfachte Interpretation der radialen Verteilungsfunktion eines Atom-Ensembles ist ein Art Wahrscheinlichkeitsverteilung der einzelnen interatomaren Abstände. Der Exponentialterm enthält zusätzlich den Glättungsparameter B, der als Temperaturfaktor (definiert die Bewegung der Atome) betrachtet werden kann.
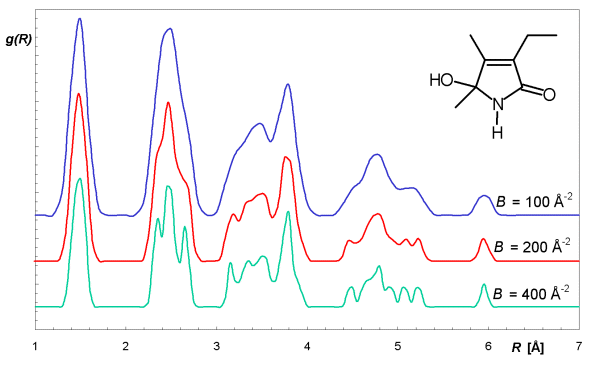
Der Einfluß des Glättungsparameters B auf den RDF Code einer Struktur (4,5-Dimethyl-3-ethyl-5-hydroxy-3-pyrrolin-2-on)
Der Einschluß charakteristischer Atom-Eigenschaften pi und pj in die Berechnung des RDF Codes macht es möglich die repräsentativen Informationen einzubinden, die für die Anforderung der jeweiligen Aufgabe nötig ist. Werden pi=pj=1 gesetzt, berücksichtigt dieser einfachste Fall nur die Struktur des Moleküls. Zum Studium eines Satzes an Liganden, die an einen Rezeptor binden, wäre es von Vorteil, Eigenschaften zu nutzen, die atomare Partialladungen beschreiben oder ihre Möglichkeit als Elektronen-Donor/Akzeptor zu fungieren.
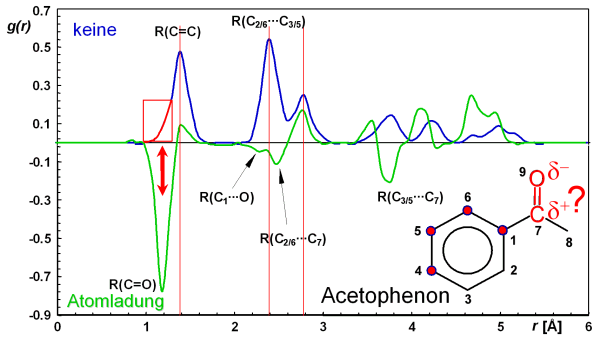
Einschluß charakteristischer Atom-Eigenschaften pi und pj in die Berechnung des RDF Codes
Der RDF Codes ist unabhängig von der Anzahl der Atome und invariant bzgl. Translation und Rotation des kompletten Moleküls.
Sowohl Regioisomere als auch unterschiedliche Konformationen können im RDF Code differenziert werden.
V. Steinhauer, J. Gasteiger, Obtaining the 3D structure from infrared spectra of organic compounds using neural networks, in Software-Entwicklung in der Chemie 11, G. Fels, V. Schubert (eds.), Gesellschaft Deutscher Chemiker, Frankfurt/Main, 1997
J. Karle, J. Chem. Inf. Comput. Sci. 1994, 34, 381-390
J. Gasteiger, J. Schuur, P. Selzer, L. Steinhauer, V. Steinhauer, Fresenius J. Anal. Chem. 1997, 359, 50-55
M.C. Hemmer, V. Steinhauer, J. Gasteiger, Vib. Spectrosc. 1999, 19, 151-164
© Prof. Dr. J. Gasteiger, Dr. Th. Engel, CCC Univ.
Erlangen, Wed Jun 9 12:55:24 2004 GMT
 BMBF-Leitprojekt
Vernetztes Studium - Chemie BMBF-Leitprojekt
Vernetztes Studium - Chemie
|


 BMBF-Leitprojekt
Vernetztes Studium - Chemie
BMBF-Leitprojekt
Vernetztes Studium - Chemie


